Introductory statistics with R: chapter 10
이번 챕터에서는 advanced data handling에 대해 배운다.
* Recoding variables
cut() function
양적인 data를 명목변수(factor)로 바꿔주는 함수. 첫번째로 data, 두번째로 breakpoint vector를 지정해준다.
interval에 포함되지 못한 자료들은 NA로 처리된다.

right=T/F argument: default는 T로, interval이 좌측으로는 <, 우측으로는 <= 로 구성된다.
include.lowest=T/F: default는 F로, T로 설정해야 1번째 interval의 좌측을 <=로 닫아준다.
right=T, include.lowest=T 를 설정해야 양 끝단의 interval이 모두 포함된다.
특정 숫자가 아닌, 동일한 크기의 그룹으로 나누기 위해선 quantile function을 함께 이용할 수 있다.


보통 quantile을 이용해 자른 것들은 결과가 예쁘지 않으므로 1st, 2cnd와 같이 levels 이름을 바꿔주는 것이 좋다.

Hmisc package에 있는 cut2 function은 이런 것들을 좀 더 simplify해서 포함하고 있다고 한다.
* Manipulating factor levels
factor를 지정할 때, levels와 labels를 구분하는 것이 중요하다. Levels는 coding되어있는 벡터를 가리키며, 여기에 해당하는 label의 factor로 코딩된다. 즉, levels가 input이고 labels가 output에 해당된다고 볼 수 있음.
levels를 따로 지정하지 않으면, 오름차순으로 sorted되어 (혹은, alphabetical하게) 부적절할 수 있음.

또한 levels를 따로 지정하지 않으면 data에 언급되지 않은 level은 포함되지 않는다.
아래는 4개로 나눠진 그룹을 3개로 묶는 과정이다.

* Working with dates
"Date"라는 클래스가 존재해서 복잡한 날짜 다루는 것을 도와준다. as.Date() 함수를 이용해 string으로 된 날짜를 date class로 변환할 수 있다.


** Date class에서 사용가능한 문자열 서식지정자는 다음과 같다.

Date class는 내부적으로, 기준 시점(epoch, 1970년 1월 1일)에서 떨어진 일수를 카운트한 숫자값이다. 따라서 class는 숫자값은 아니지만, Date class 간 사칙연산이 가능하며 summary를 해도 quantile 값을 얻을 수 있다.

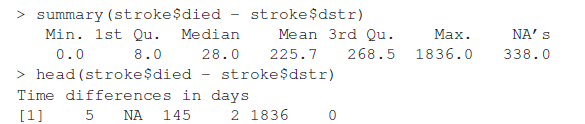
Data 간 뺄셈을 한 object의 data type은 "difftime"이다. difftime은 unit에 따라 days, hours, seconds 등 다르게 계산될 수 있으므로 as.numeric으로, unit을 명시한 뒤 숫자로 변환하여 계산하는 것이 더 추천된다고 함.

units를 "years"로 하지 않은 이유는, units의 최대 단위는 "weeks" 이기 때문. Epidemiological year를 계산하기 위해서는 365가 아닌 365.25로 나눠주는데, 0.25는 leap years(윤년)을 보정하기 위한 수치라고 한다.
stroke object에 end와 dead라는 항목을 추가한다. end는 사망시점이 있다면 그 시점을, 사망하지 않았다면(본 데이터 상에는 NA로 표시되어 있음) 마지막 follow up인 1996.01.01을 입력하는 것이다. min(), max() 함수는 입력받은 값 중 최대/최소값을 출력해주는 함수이고, pmin() / pmax()는 입력된 벡터와 "parallel"인 값들을 비교해서 single vector를 return 해주는 함수이다. dead는 died가 NA라면 FALSE이다. 따라서 !is.na(died)를 사용하고, died가 NA가 아니라면 마지막 follow up인 1996-1-1보다 작아야 관찰기간 내 사망으로 볼 수 있다. 따라서 as.Date("1996-1-1")과 비교를 하였다.
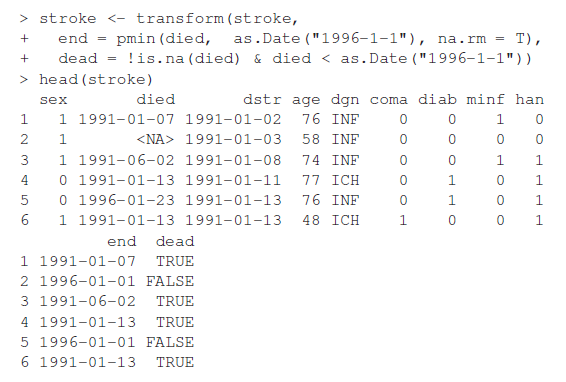
** transform() 함수에서는, 아직 지정되지 않은 변수에 대해 사용할 수 없다. 즉, 하나의 호출 내에서 만들어진 변수는 해당 호출 내에서 다시 사용할 수 없다. 이를 원하는 경우에는 within 함수를 사용할 수 있다고 한다.
* Further time classes
Date 이외에 R 내에 내장된 시간 함수는 POSIXct 라는 class가 있다. POSIX 기준에 맞춘 calendar time으로, date와 비교해서 다른 점은 day가 아닌 second를 단위로 한다는 것이며, 보단 POSIXlt(local time)은 year, month, day of month, hours, minutes, seconds 등 다양한 구조를 가진다는 점이다.
* Recoding multiple variables
앞에서 다룬 예제에서는 여러 번의 호출을 통해 데이터를 수정하였다. 하지만 Data frame은 근본적으로 list의 일종이므로, lapply를 이용하면 동일한 조작을 한번에 할 수 있다.

* Conditional calculations
ifelse() function을 이용하면 특정 변수의 조건에 따라 다른 값들을 적용할 수 있다. 아래 에시를 보면, event = !is.na(died)를 이용해 사망날짜가 있는 경우 TRUE, NA인 경우(사망 안함) FALSE가 표시되도록 만들었다. 그리고 ifelse function을 이용해 event의 값이 TRUE, 즉 사망한 날짜가 존재하는 경우 obstime에 died-dstr값을 넣도록 하였고 만약 event값이 FALSE라면 그 뒤에 있는 as.Date("1996-1-1")-dstr 값, 즉 마지막 관측일에서 dstr을 뺀 시간을 넣도록 하였다. ifelse() function의 경우, ifelse(조건, 참인경우 시행할 구문, 거짓일 경우 시행할 경우0 로 구성된다.

* Combining and restructuring data frames
* Appending frames

이 부분에서는 data frame을 vertically, or horizontally 합치는 방법을 알아보자.

juul의 예시를 들어서 보면, juul에서 sex에 따라 남/녀로 자료를 분류한다. 이 때 sex==2에 필요 없는 testvol, sex는 제외한 상태로 subset을 만든다. sex==1도 마찬가지로 만든다. 이후 sex는 범주형변수로 변환해 F, M으로 각각 추가하고 testvol, menarche는 NA로 일괄 입력한다. 이후 rbind를 통해 두 data frame을 합칠 수 있다. rbind()를 이용할 때, 각 자료는 column name에 따라 정렬되어 합쳐지며, column name의 순서가 다를 경우 rbind의 앞부분에 언급된 object의 순서를 따라간다. 그리고 sex에 대해, 각 object 별로 F, M만 할당한 상태로 rbind를 했더니 factor는 이 모든 요소를 고려해서 M, F 두 개로 변경되었다.
* Merging data frames
위에 rbind로 vertical stacking을 했던 것과 마찬가지로 cbind로 column들을 붙일 수 있다. 하지만 순서가 잘못 맞거나 데이터에 누락이 있는 경우 등 위험할 수 있다. 이 때 merge function을 사용해볼 수 있다.
merge() function은 동일한 이름을 가진 것들끼리 합쳐준다.
merge(dfx, dfy, by="ID") 이런 식으로 사용할 수 있음.
merge() function을 이해할 수 있는 예제
df1, df2를 다음과 같이 설정한 뒤, df1의 name - df2의 product를 짝 맞춰, 동일한 key값을 지닌 것들을 동일한 것으로 생각해 merge를 시행하려 한다. 이 때, by.x는 x의 키값이 들어가는 항목, by.y는 y의 키값이 들어가는 항목을 지정해주면 된다. 여러 항목을 시행할 경우 벡터를 사용해도 됨.
merge(df1, f2, by.x='name', by.y='product')를 시행했더니 b,c만 출력되었다. (그리고 df1의 column인 'name'으로 합쳐졌다. merge 역시 앞에 언급된 object의 명칭을 이어받는 방식) 왜냐하면 df1, df2에 공통적으로 존재하는 것이 b, c 밖에 없기 때문이다. df1에 있는 요소들을 기준으로 해서 합치기 위해선 all.x=T를 설정해야 한다. 그러면 x의 모든 요소들에 대해 merge 된다. 반대로 df2의 요소를 기준으로 하기 위해선 all.y=T를, 그리고 모든 요소를 빠짐없이 하여 합치기 위해선 all=T로 설정해야 한다.

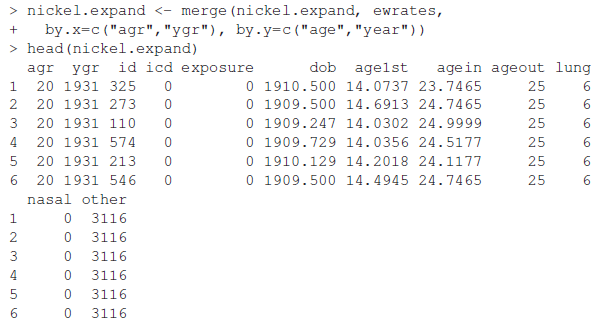
책의 예제에서는 ISwR 내 데이터셋인 nickel과 ewrates를 합치는 과정을 보여준다. 먼저 이 두 데이터의 특성을 비교해보자. head() 함수를 써서 나타낸 겂은 다음과 같다.

nickel의 agein과 ewrates의 age, 그리고 nickel의 dob+agein과 ewrates의 year를 합칠 생각이다. 자료의 특성을 보면, ewrates에서 age는 5세 간격으로 끊겨있다. 즉, 5살 간격 구간으로 바꿔줘야 한다. 10-14세는 10세로, 15-19세는 15세로 바꿔주는 조작을 할 수 있다. 그리고 year는 5의 배수 +1의 모양을 하고 있으므로 이 역시 마찬가지로 바꿔줄 수 있다.

정수를 반올림하기 위해서는 round()라는 함수를 쓸 수 있다. 본문에서 사용한 trunc함수는 "버림" 함수이다. 나이를 5로 나눈 뒤 trunc()해서 정수를 만들고, 이를 다시 5를 곱해서 복구한다. 이런 방식으로 10-14세는 10세로, 15-19세는 15세로 변경할 수 있다. 14세에 대해 계산을 해보면, 14/5=2.8, trunc(2.8)=2, 2*5=10.. ygr에서 -1을 하고 후에 +1을 해주는 이유는, 5의배수 +1의 형태로 만들어주기 위해서이다.
이제 동일한 키값들을 생성했다면 merge()해보자.

nickey의 "agr", "ygr"이 ewrates의 "age", "year"에 대응되도록 합쳤다. 동일한 agr, ygr를 갖는 값이 nickel에는 여러 가지가 있었다. 이는 다른 id로 구분된다. ewrates의 다른 column인 nasal, lung 등을 보면, 동일한 키 값을 갖는 여러 요소에 대해서는 하나의 항목이 복사됨을 알 수 있다.
* Reshaping data frames
한 피험자에 대해 여러 시점에서 지속적인 측정을 하는 시험을 했다고 하자. 항암치료를 받는 사람에서 alkaline phosphate level을 3개월 단위로 측정하는 시험의 예를 들어보자. 이 자료를 표시하는 방법에는 2 가지 포멧이 있다: "wide", "long" format.
wide의 경우, 각 unit이 한 row를 차지하며 column에는 3개월 단위로 측정한 값들을 넣는다. 따라서 여러 column으로 data가 구성되게 되며, unit의 숫자가 즉 row의 숫자가 된다. 여러 column을 사용하여 이를 "wide"라고 한다.

또는, 한 환자에 대해 측정 시기(0, 3, 6, 9 개월 등)를 하나의 column으로 하여 길게 나열할 수도 있다.

이를 "long"이라고 한다.
reshape() function을 이용해 이 둘을 필요에 따라 상호변환할 수 있다. 책 본문의 예제를 따라가보자.

alkfos를 a2 object에 할당했고, a2의 이름을 변경하였다. sub() 함수란, 하나의 단어를 다른 단어로 교체한 뒤 교체된 object를 반환하는 함수이다. sub(pattern, replacement, object)의 순서로 입력하면 된다. 위와 같이 이름을 바꿔준 이유는, reshape함수가 "time varying variables"를 찾을 수 있게 하기 위함이다. Default로, '.'를 이용해 time varying variable과 앞의 다른 문자를 구분하도록 되어있다. 따라서 위와 같은 조작을 하지 않는 경우, c 뒤의 숫자를 각각의 시기로 인식하지 못하여 아래와 같은 경고메시지가 나온다.


reshape()함수는 위와 같이 사용한다. varying은 time-varying variable이 존재하는 column의 index를 지정해준 것이다. 즉, 2-8번 column이 time varying variable이니 이를 longitudinal하게 바꿔달라는 요구이다. direction="long"이므로 수직하게 바꾸는 시행을 한다. 결과는 위와 같다. 각각의 time(0, 3, 6, .. 24)에 대해 id(1-43)의 alkphos 농도(='c'값)를 longitudinal하게 나열했다.
시간 순이 아니라 각 unit 당 시간에 따라 모아서 보고 싶다면, 아래와 같이 호출하면 된다. order(id, time) 명령을 이용하면 id를 이용해 먼저 정렬을 하고, 동일한 id 내에서 time에 따라 정렬을 한다. (정확히 말하면, 어떤 방식으로 정렬해야 하는지 그 순서를 벡터로 반환해줌). 따라서 해당 벡터를 row에 넣어 재배치하면 아래와 같아진다.

이렇게 reshape를 하고 나면, 다시 되돌리기 위해서는 단순히 reshape(a.long)만을 하면 된다. 왜냐하면 필요한 data들은 R에서 알아서 attributor에 넣어두기 때문이다. attr(a.long, "reshapeLong")을 시행해보면 아래의 attributor들을 볼 수 있다.

본문에서는 이를 배제하고, 애초에 long 한 자료가 주어졌을 경우를 상정해 이를 삭제하고 reshape을 진행한다.

na.omit은 missing data를 제거하기 위해 사용하였다.
a.long2를 wide로 바꾸기 위해 아래와 같이 세팅해주면 된다. argument 중 idvar, timevar는 각각 id variable, time variable을 의미한다. v.names argument는 time-varying variable을 의미한다.

* Per-group and per-case procedure
자료를 그룹별 처리가 필요한 상황들이 있다. 이를 하기 위해서, 자료를 그룹별로 나눈 뒤 조작을 하고, 다시 합치는 방법이 유용하다. 본문에서는 위에서 다룬 a.long object를 id별로 분류하여, 각 측정값들을 해당 피험자의 0개월 측정결과에 대한 비율로 나타내려고 한자. 이 때, id별로 데이터를 나누기 위해 split()함수를 사용한다. split() 을 사용하면 명목변수 (factor)에 따라 자료를 분류해 list로 만들어준다. split(data, factor) 이런 식으로 사용하면 된다.

이후 lapply로 각 list의 요소들에 function(x) x/x[1]을 적용해준다. 해당 vector의 첫번째 값으로 해당 벡터 전체를 나눠주게 되면 0개월차 값에 대한 비율이 되기 때문이다.

원하는 조작이 완료되었다면, unsplit()을 이용해 다시 되돌릴 수 있다. unsplit을 이용하면 id에 따라 해당값을 나열한 벡터를 얻을 수 있으므로, a.long에 새로운 column을 생성해 집어넣을 수 있다.

ave()
이 split - lapply - unsplit을 한번에 해주는 function이 있다. 바로 ave() function이다.

혹은, 아래와 같이 lapply() 함수와 transform() 함수를 사용할 수도 있다.

* Time splitting
Ad hoc programming: It basically means writing some quick and dirty code without the intention of reuse.
Ad hoc programming이란, 처음부터 systematic하게 접근하는 것이 아닌 해당 문제만을 해결하기 위해 다소 즉흥적이고 빠른 방식으로 이뤄지는 프로그래밍을 말한다. 이번 chapter에서는 ad hoc programming의 방식으로 nickel data를 subdivision으로 나누고, 이를 변수를 이용해 일반화한 함수로 만드는 예제를 살펴본다.

먼저 nickel의 data를 살펴보면, id는 환자의 id, ICD는 international classification of disease에 의한 사망 원인의 질병코드, 그리고 dob는 date of birth, age1st 는 첫 노출 나이, age in 및 out은 follow up 시작 및 종료 시 나이를 의미한다.
이 데이터는 니켈이 노출된 환자들이 어떤 사인으로 사망했는지는 (특히 nasal, lung cancer) 조사한 것이다. 암은 오랜 기간에 걸쳐 발생하고 사망을 초래하는 질환이므로, 장기간에 걸친 환자 f/u을 5년 주기로 나눠서 분석할 필요가 있다.
5년 간격으로 나이를 자르고, 환자를 sub-individual로 나누는 작업은 복잡하기 때문에 60-65세 사이의 환자를 처리하는 예시 (nickel60)을 먼저 만들어본 뒤 이를 일반화해보자.
먼저, 환자의 agein이 60세보다 어리다면, 이 데이터는 60-65세의 subdata이므로 최소나이를 60세로 맞춘다. 그리고 65세보다 많다면 최대나이를 65세로 맞춘다. entry, exit은 각각 시작, 끝 연령이 된다. 60-65세 사이에 agein, ageout한 피험자가 있다면 고유의 값을 갖겠지만 60-65세를 포함하여 그보다 큰 범위에 agein, out한 피험자는 60-65를 갖게 된다.

valid는 entry < exit을 비교한 논리연산자 벡터로 만든다. entry<exit의 이유는, 만약 환자가 60세 이전에 혹은 65세 이후에 agein/out을 했다면, entry > exit이 된다. (몇가지 예로 직접 해보면 이유를 알 수 있다). 따라서 올바른 배열로 entry와 exit의 크기가 존재하는 것만이 60-65세 연령구간을 포함하는 valid한 요소임을 생각한다. 그리고 entry와 exit을 전체 data에서 valid한 요소만 추린 값으로 만든다.

cens는, 만약 ageout이 65세보다 크다면 환자는 이 구간에서 사망하지 않은 것이다. 이 구간을 cens로 표시하고, ICD는 질병 사인을 의미하므로 0을 넣기로 한다.

nickel60은 nickel에서 valid한 것들만 모아서, cens에 해당되는 항목은 ICD를 0으로 수정한 뒤 agein은 entry로, ageout은 exit으로 입력을 한다. 60-65 구간이므로 agr은 60세, ygr은 위의 공식을 참조해서 만든다.

위의 과정을 일반화하면, trim이라는 function에 start를 변수로 받아들여 start와 end를 5 차이 나는 값으로 둔 뒤, 각각 entry와 exit, valid, cens 등 완전히 동일한 과정을 거친 뒤 만들어진 result data frame을 반환하도록 만든다.
위와 같이 함수를 만들었다면, trim(60)만 시행해도 위의 복잡한 과정과 동일한 결과를 얻을 수 있다.
우리는 이제 20세부터 95세까지 5세 간격으로 끊어서 이 trim을 적용하려고 한다. 그리고 이렇게 만들어진 data frame 들을 rbind하여 각 sub-individual로 구성된 새로운 data frame을 만드려고 한다.

위의 do call 함수를 이용하면 간단하게 사용 가능하며, do.call은 입력받은 리스트 모두에 해당 함수를 "한번" 적용해주는 것이다. 이를 풀어서 쓰자면 아래와 같다.

id가 4인 피험자에 대한 자료를 꺼내보면 아래와 같이 5세 간격의 sub-individual로 나뉘었음을 알 수 있다.

마지막으로 위에서 했던 것과 마찬가지로 ewrates data를 merge해서 합쳐주면 완성.